Das Gehirn besteht aus einem riesigen Netzwerk von Neuronen. Das Verständnis, wie Zellen in solchen Netzwerken interagieren, ist unerlässlich, um medizinische Hilfsmittel zur Diagnostik und Behandlung von Hirnschädigungen zu entwickeln und zu verbessern. So entwickeln Wissenschaftler beispielsweise elektronische Prothesen mit dem Ziel, die Funktionsfähigkeit des geschädigten neuronalen Gewebes wiederherzustellen – um jedoch die Stimulationsgenauigkeit der Prothesen verbessern zu können, ist ein tieferes Verständnis des geschädigten Systems erforderlich. Um die Vielzahl der Mechanismen zur Erzeugung neuronaler Aktivität – sowohl die Prozesse in den einzelnen Neuronen als auch die Interaktion in den Neuronennetzen – zu verstehen, können Rechenmodelle verwendet werden. Die Identifizierung geeigneter Modelle (d. h. Modelle, deren Output mit den Messdaten übereinstimmt) hat sich jedoch bisher oft als sehr schwierig erwiesen, insbesondere für komplexe, mechanistische Modelle mit zahlreichen Parametern. In zwei kürzlich erschienenen Arbeiten haben wir eine neue Methode zur Identifizierung geeigneter Modelle entwickelt und auf ein Rechenmodell für Netzhautprothesen (d. h. Implantate zur Wiederherstellung des Sehvermögens bei Blinden) angewendet.
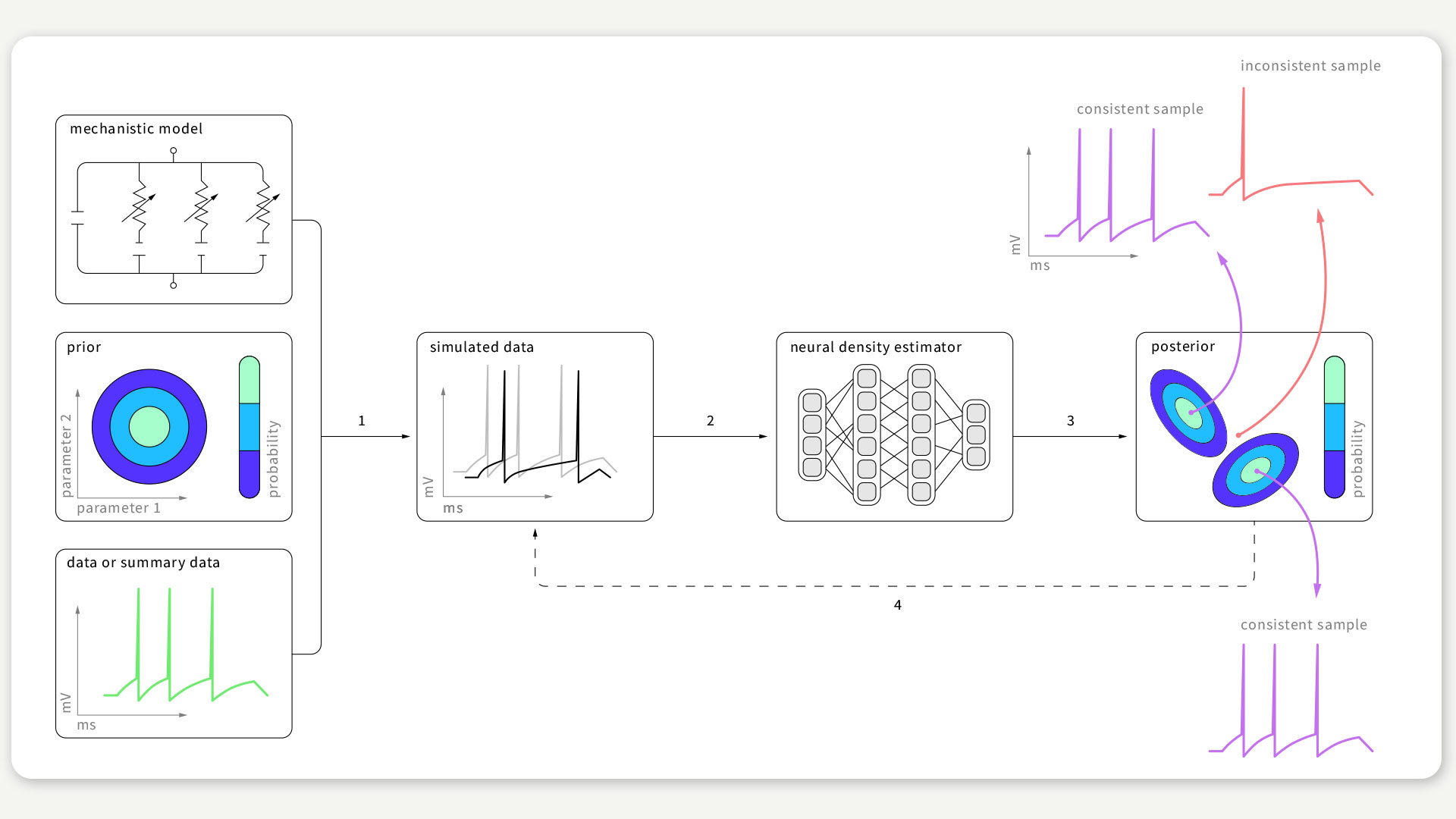
ABBILDUNG 1 / Der Algorithmus „Sequential Neural Posterior Estimation“ (SNPE) soll Modelle finden, die mit den Messdaten kompatibel sind. Dies erfordert drei Inputs: Ein Modell (d. h. ein Computercode zur Simulation von Daten mit Hilfe von Parametern), Vorwissen bzw. Rahmenbedingungen für die Parameter und empirische Daten. SNPE führt Simulationen für verschiedene Parameterwerte durch und trainiert ein künstliches neuronales Netz, um jedes Simulationsergebnis auf eine Reihe möglicher Parameter abzubilden. Nach dem Training mit bekannten Modellparametern anhand der simulierten Daten liefert SNPE die gesamte Bandbreite an Parametern, die mit den empirischen Daten und dem Vorwissen übereinstimmen. Abbildung nach Gonçalves et al., 2020.
Von Versuch und Irrtum zu einer algorithmischen Identifizierung geeigneter Modelle
In einer Veröffentlichung mit den Hauptautoren Pedro Gonçalves, Jan-Matthis Lueckmann und Michael Deistler aus der Arbeitsgruppe von Jakob Macke haben wir uns mit der Frage beschäftigt, wie man geeignete Modelle am besten bestimmen kann. Bisher gab es dafür nur begrenzte Möglichkeiten: Entweder wurden vorhandene Modelle in mühevoller Arbeit so lange modifiziert, bis sie die Messdaten erklären konnten, oder es wurde versucht, Modelle durch eine heuristische Vorgehensweise durch exzessives Ausprobieren mit den Messdaten in Übereinstimmung zu bringen – d.h. durch Testung so vieler Modelle, wie es die vorhandenen Datenverarbeitungsressourcen erlauben. Entweder sind diese Ansätze nicht systematisch und unterliegen subjektiven Kriterien, oder sie sind ineffizient und daher begrenzt durch die Komplexität des Modells.
Um diese Einschränkungen zu überwinden, haben wir einen Algorithmus entwickelt, der geeignete Modelle automatisch anhand der experimentell erhobenen Daten bestimmt. Entscheidend bei diesem Ansatz ist die „Bayessche Inferenz“: Mit Hilfe der gemessenen Daten und der Modellsimulationen identifiziert der Algorithmus sämtliche geeignete Modelle, die zu den Messdaten passen.
Bemerkenswert hierbei ist, dass dieser neue Ansatz keinerlei Kenntnisse der genauen Funktionsweise des Modells erfordert, so dass er auf verschiedene Modellarten mit ganz unterschiedlicher Komplexität anwendbar ist. Wir haben die Effizienz des Algorithmus anhand mehrerer kanonischer Modelle aus der Neurowissenschaft belegt und gehen davon aus, dass er auch in anderen Teilbereichen der biomedizinischen Forschung eingesetzt werden kann.
Anwendung: Ein Modell in der retinalen Prothetik, um das Sehvermögen wiederherzustellen
In der Arbeitsgruppe von Philipp Berens haben Jonathan Oesterle, Christian Behrens und andere diesen Algorithmus verwendet, um Modellparameter für drei Neuronenarten der Retina (Netzhaut) abzuleiten. Neben der Bestimmung der Parameter bestand ein Hauptziel der Studie darin, die Kluft zwischen der theoretischen Modellierung und der medizinischen Anwendung zu überbrücken. Zu diesem Zweck haben wir ein Modell für die externe elektrische Stimulation retinaler Neuronen geschaffen, das die retinale Stimulation simulieren soll, die in Neuroprothesen für die Netzhaut zur Anwendung kommt. Wir haben verschiedene elektrische Stimuli untersucht und dabei nach Reizen gesucht, die entweder On- oder Off-Bipolarzellen (bezogen auf das unterschiedliche Verhalten bei Lichteinfall auf die vorgeschalteten Rezeptorzellen) gezielt stimulieren, da auf diese Weise visuelle Szenen besser in elektrische Signale kodiert werden könnten. Dies stellt zur Zeit eines der Hauptprobleme bei Retina-Implantaten dar.
Für einen solchen modellgetriebenen Ansatz ist die sorgfältige Auswahl der Modellparameter von entscheidender Bedeutung. Deshalb wurden die Parameter für die modellierten retinalen Neuronen zunächst basierend auf Informationen aus der Literatur geschätzt und eingeschränkt. Da zu wenige Daten verfügbar waren und diese die Modellparameter nicht ausreichend einschränkten, haben wir mit Hilfe des SNPE-Algorithmus die Zellparameter weiter optimiert und Zellmodelle erhalten, die mit den Antworten auf Lichtreize übereinstimmen, die zuvor mit der Zwei-Photonen-Mikroskopie erfasst worden waren. Mit Hilfe des Algorithmus wurden außerdem die Parameter der elektrischen Stimulation abgeleitet.
Durch die Kombination der Zellmodelle mit dem Modell für die elektrische Stimulation konnten wir die Auswirkungen unterschiedlicher Stimulationsansätze in silico (in der Computersimulation) untersuchen. Nach der Validierung unserer Simulationen durch verfügbare Daten zu Reizschwellen für verschiedene Reizsituationen testeten wir eine Vielzahl von elektrischen Reizen für die selektive Stimulation von On- bzw. Off-Bipolarzellen. Unser Modell führt zu der Annahme, dass triphasische Ströme möglicherweise in der Lage sind, die Off-Zelle aufgrund ihrer Exprimation der Calciumkanäle des T-Typs gezielt reizen zu können. Diese Kanäle öffnen sich bei relativ starken, aber kurzzeitigen Erhöhungen des Reizstromes, was wiederum zu einer Calcium-aktivierten Neurotransmitterfreisetzung führt. Andererseits konnten wir keine Stimuli finden, die ausschließlich On-Zellen reizen.
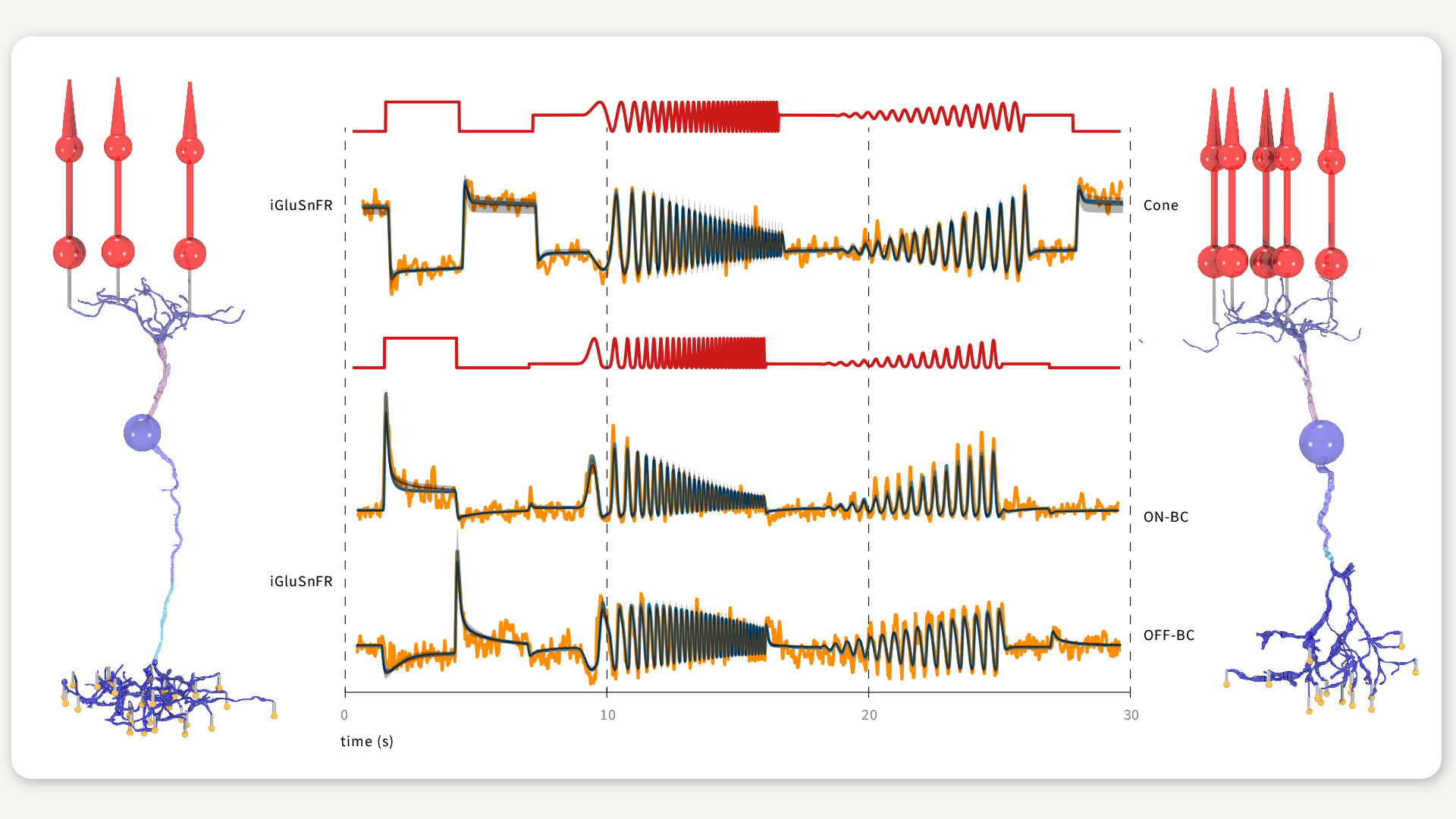
ABBILDUNG 2 / PARAMETERINFERENZ FÜR MODELLE VON NERTZHAUTZELLEN In der Arbeitsgruppe von Philipp Berens erfolgte die Ableitung der Parameter für eine Zapfenzelle und eine On-Bipolarzelle (linke Zelle) sowie Off-Bipolarzelle (rechte Zelle), wobei die Zellen Input von drei bzw. fünf Zapfen (rote Zellen) erhielten. Die Outputs des Modells (blau und schwarz) wurden mit experimentell erfassten Antworten (orange) auf einfache Lichtreize (rote Kurve) abgeglichen, die den experimentellen Bedingungen entsprachen. Abbildung nach Oesterle et al., 2020.
Schlussfolgerungen
Die beiden beschriebenen Studien zeigen, wie die Entwicklung eines Verfahrens und seine Anwendung auf anspruchsvolle wissenschaftliche Fragestellungen Hand in Hand gehen und einander ergänzen können. Wir glauben, dass diese Erkenntnisse nicht nur einen wichtigen Beitrag zur Entwicklung effizienter Neuroprothesen für Blinde leisten, sondern dass sie auch den Nutzen und die Herausforderungen unseres Inferenz-Werkzeugs zeigen. Künftig könnte dieses Werkzeug in einem breiten Spektrum von computergestützten Untersuchungen sowohl in der Neurowissenschaft also auch in anderen Bereichen der Biologie angewendet werden und so dazu beitragen, die Kluft zwischen datengesteuerten und theoriegeleiteten Ansätzen zu überbrücken.
Originalpublikationen:
Pedro J Gonçalves, Jan-Matthis Lueckmann, Michael Deistler et al. Training deep neural density estimators to identify mechanistic models of neural dynamics. eLife 2020;9:e54997.
Text: Michael Deistler, Jonathan Oesterle
Mitwirkung bei der Übersetzung ins Deutsche: Chris Richardson
Titelillustration: Franz-Georg Stämmele

So verbessert maschinelles Lernen die 3D-Bodenkartierung


Kommentare