Im Wettstreit darüber, wer bessere Sprachmodelle trainiert, die Bilder erzeugen und mit den Usern in Dialog treten können, geben Technologieunternehmen jeden Monat Milliarden von Dollar aus und verbrauchen Millionen von Haushaltsstunden an Energie [1]. Heutige KI-Systeme verwenden neuronale Netzwerkarchitekturen wie etwa Transformers, die oft als „Black Boxes“ mit Milliarden von Parametern arbeiten.
Während die Optimierung dieser KI-Systeme viel Geld und Energie verbraucht, hat die Natur im Laufe der Zeit elegantere Lösungen für das Design neuronaler Netze entwickelt. So stellt Drosophila melanogaster, die genügsame Fruchtfliege mit einem Gehirn so groß wie ein Mohnsamen, aber einer bemerkenswerten Recheneffizienz, jedes heutige künstliche Netzwerk in den Schatten.
Vom Navigieren in komplexen Flugmustern über das Erkennen von Feinden bis hin zur Partnersuche – dieses winzige Lebewesen überlebt problemlos in der realen Welt. Das Gehirn der Fruchtfliege führt all diese parallelen Berechnungen mit unübertroffener Effizienz aus, benötigt dafür aber nur ungefähr 130.000 Neuronen. Diese Berechnungen werden mit einer geschätzten Leistung von 120 Nanowatt durchgeführt – das ist achtmal weniger als der Stromverbrauch einer Quarzuhr, die nur die Zeit anzeigen kann. Diese außergewöhnliche Effizienz, die in Jahrmillionen der Evolution perfektioniert wurde, ermöglicht komplexes Verhalten bei einem Stromverbrauch, der gegen null geht.
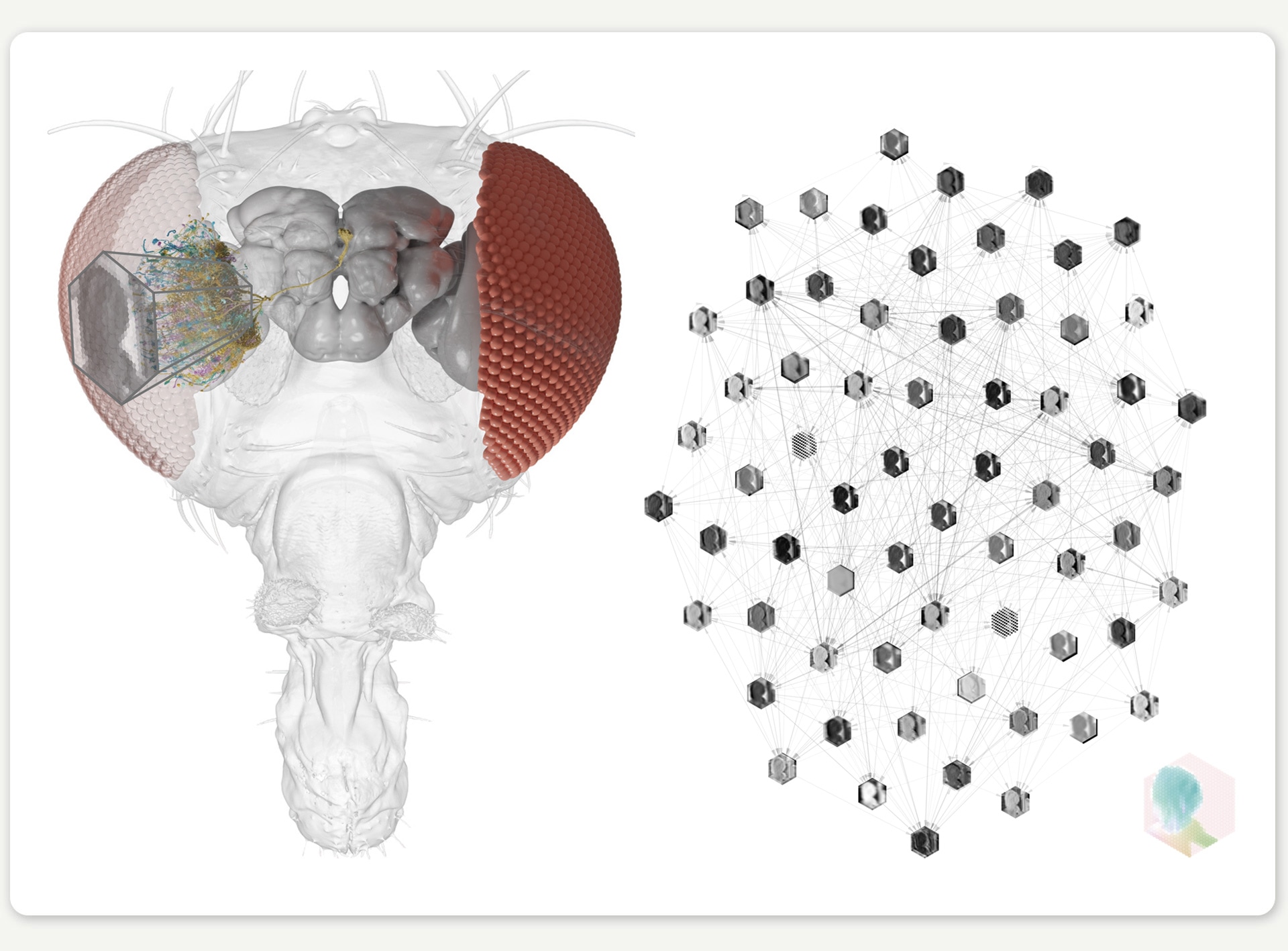
Abbildung 1: Licht fällt in das Facettenauge der Fliege und veranlasst die Fotorezeptoren, elektrische Signale durch ein komplexes Netzwerk von Neuronen zu senden, wodurch die Fliege Bewegungen wahrnehmen kann. Neuronen von Fruchtfliegen erkennen Bewegungen innerhalb von 30 Millisekunden. Diese können ein Fluchtmanöver in der Hälfte der Zeit einleiten, die ein Mensch zum Blinzeln braucht.
Wichtige wissenschaftliche Entdeckungen rund um Drosophila
Seit fast einem Jahrhundert ist dieses Insekt für eine Vielzahl wissenschaftlicher Entdeckungen in der Biologie verantwortlich, von denen einige mit dem Nobelpreis ausgezeichnet wurden. Drosophila wurde zum Beispiel als Modellsystem zur Identifizierung von Genen genutzt, welche den zirkadianen Rhythmus und die Entwicklung steuern. Der schnelle Fortpflanzungszyklus – in nur 10 Tage vom Ei zum erwachsenen Tier – ermöglicht es den Forschenden, mehrere Generationen innerhalb von Wochen anstatt von Jahren zu untersuchen. Genetische Werkzeuge wie das GAL4-UAS-System erlauben eine präzise Kontrolle des Phänotyps, einschließlich der Expression spezifischer Eigenschaften von Neuronen. Messtechniken, die von der Elektrophysiologie bis zur Kalzium-Bildgebung reichen, ermöglichen es, die neuronale Aktivität in der gesamten Fliege zu erfassen. In Kombination mit umfangreichen Verhaltensaufzeichnungen macht dies die Fruchtfliege zu einem idealen Versuchsobjekt, um Ursache und Wirkung von biologischer Intelligenz zu verstehen.
Aufbauend auf dieser umfangreichen wissenschaftlichen Grundlage konnten mehrere Forschungsteams (insbesondere das FlyEM-Projekt am Janelia Research Campus in Virginia und das FlyWire-Konsortium) das gesamte Konnektom der Fliege kartieren und so ein umfassendes Diagramm aller Neuronen und neuronalen Verbindungen in ihrem Gehirn erstellen. Diese digitalisierte Darstellung der Gehirnstruktur liefert eine vollständige Karte der elektrischen Signalübertragung zwischen Neuronen und ermöglicht einzigartige Einblicke in die Informationsverarbeitung eines biologischen neuronalen Netzwerks.
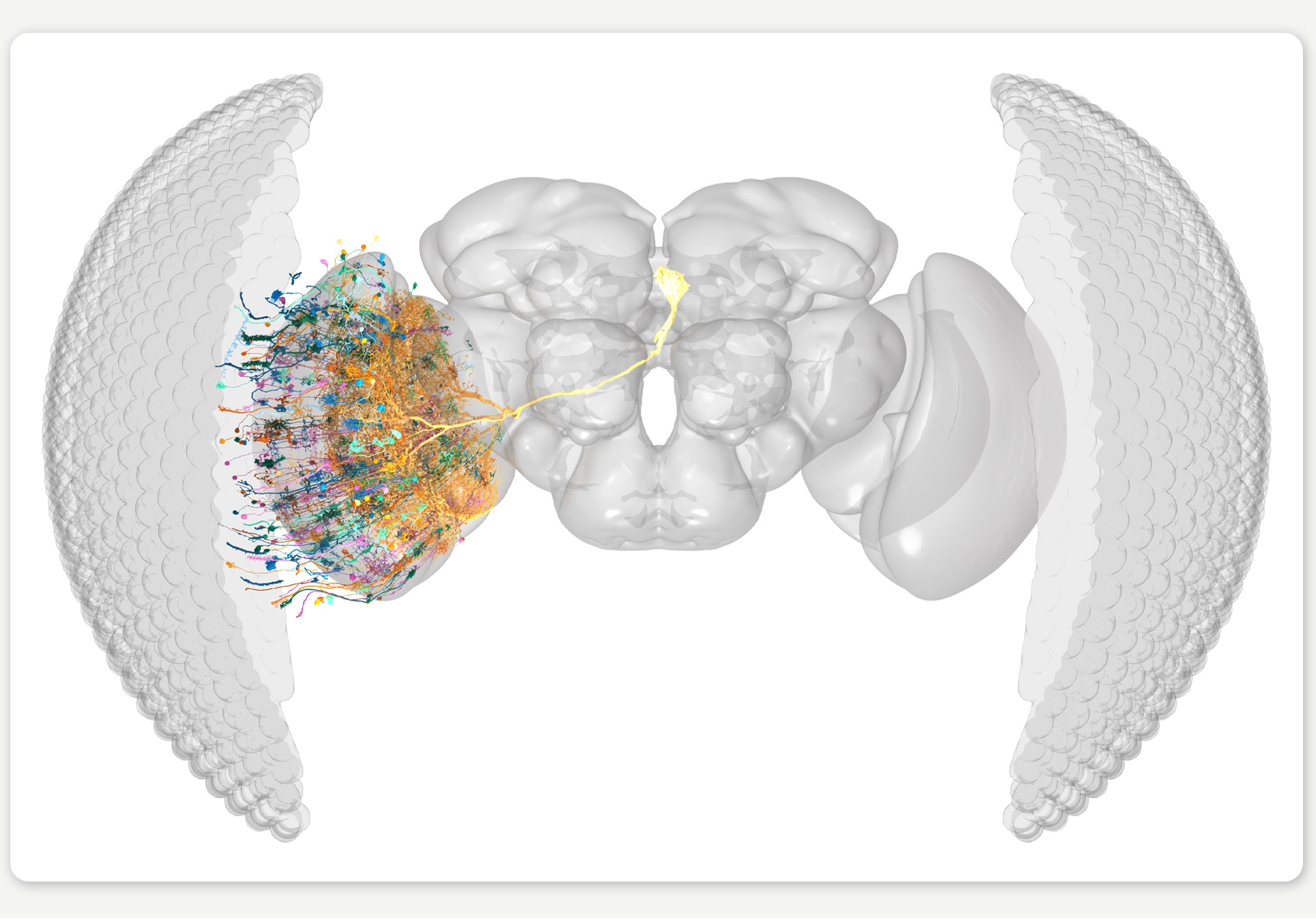
Abbildung 2: Darstellung des Konnektoms des rechten Auges, die zur Veranschaulichung nur eine kleine Anzahl von Neuronen zeigt. ©SIWANOWICZ, I. & LOESCHE, F. / HHMI JANELIA RESEARCH CAMPUS
Verstehen, wie ein Konnektom Daten verarbeitet
Ein Konnektom zu haben ist die eine Sache, zu verstehen, wie es Daten verarbeitet, eine ganz andere. An dieser Stelle setzt unsere Forschung an, ein gemeinsames Projekt der Arbeitsgruppen von Jakob Macke (Universität Tübingen) und Srinivas Turaga (HHMI Janelia Research Campus Ashburn, Virginia). In unserer Studie, welche kürzlich in Nature veröffentlicht wurde, haben wir ein neuronales Netzwerkmodell entwickelt, in dem jedes künstliche Neuron einem echten Neuron im Gehirn der Fruchtfliege zugeordnet wird. Im Gegensatz zu den meisten künstlichen neuronalen Netzwerkmodellen, die Milliarden von Parametern beinhalten, verwendet unser Konnektom-basiertes Modell nur 735 sorgfältig zugeordnete Parameter. Da wir zu Beginn nicht abschätzen konnten, welche Parameterwerte genau benötigt werden, haben wir mit einer ersten Schätzung begonnen, um das Modell dann für verhaltensrelevante Aufgaben zu optimieren. Da die Bewegungserkennung für das Überleben der Fruchtfliegen entscheidend ist, trainierten wir unser Modell dahingehend, optischen Fluss in naturalistischen Videos zu erkennen. Durch dieses Training ahmen die dynamischen Berechnungen des Modells eine der wichtigsten visuellen Fähigkeiten der Fliege nach.
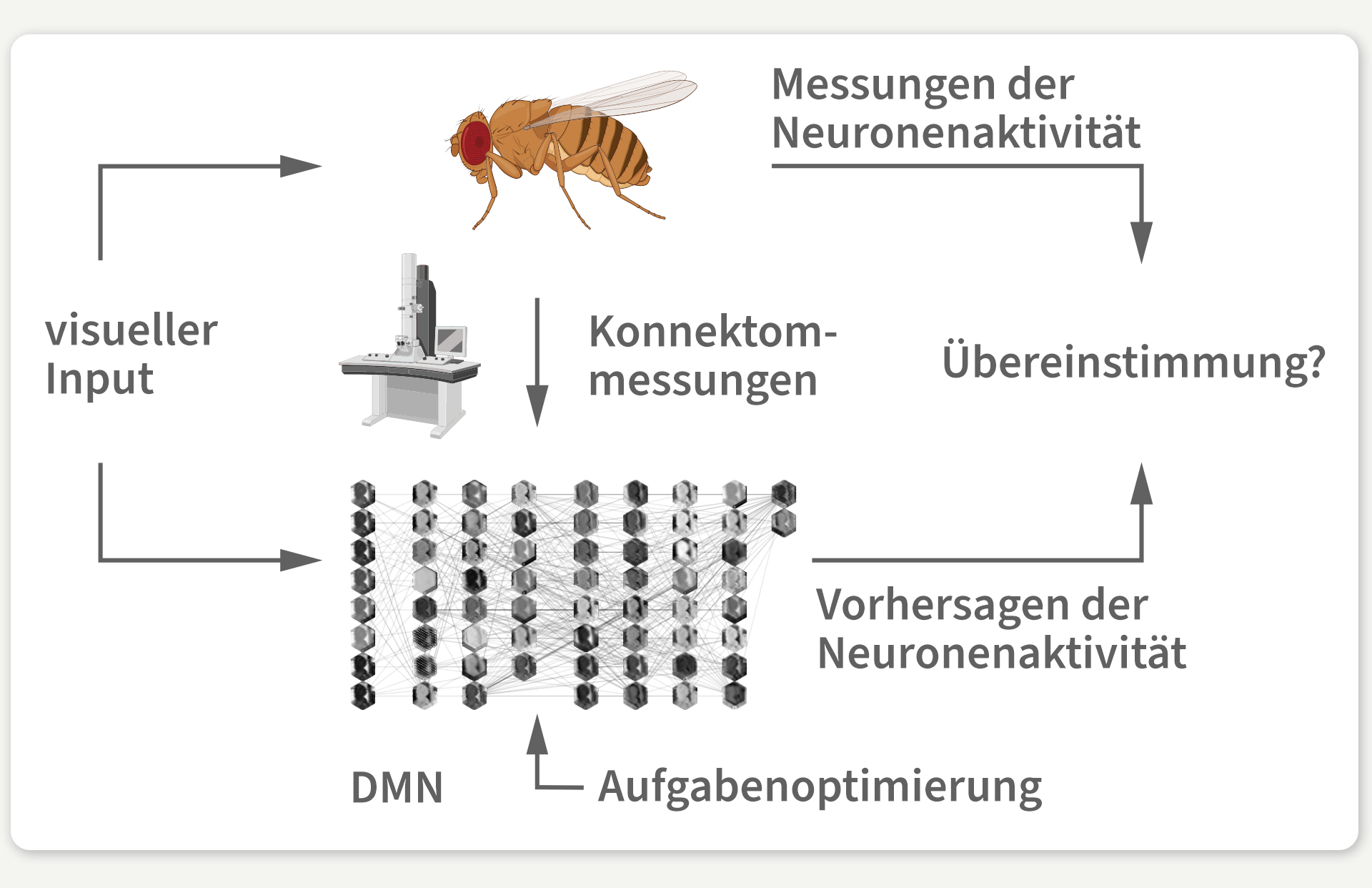
Abbildung 3: Wir verwenden das Konnektom, um eine detaillierte, tiefgreifende mechanistische Netzwerksimulation des visuellen Systems der Fliege zu erstellen, mit der sich die neuronale Aktivität in lebenden Gehirnen vorhersagen lässt.
Unser Modell wird nur durch die Zuordnung von künstlichen zu echten Gehirnneuronen, dem dynamischen Neuronenmodell und der Bewegungserkennung gesteuert. Wir konnten nicht sicher sein, ob die Aktivität der einzelnen Neuronen im Modell mit den tatsächlichen Messungen im Tiergehirn übereinstimmen würde. Deshalb haben wir die neuronalen Antworten unseres Modells auf Stimuli aus Laborexperimenten quantifiziert, um Prognosen unseres eigenen Modells mit tatsächlichen Messungen der neuronalen Aktivität einzelner Neuronen abzugleichen.
Praktischerweise ähnelt bei den Fruchtfliegen ein Gehirn dem anderen sehr – und jetzt auch unserem Modell. Wir haben festgestellt, dass die Aktivität jedes künstlichen Neurons in unserem Modell als Reaktion auf die Laborstimuli mit der Aktivität seines echten biologischen Gegenstücks übereinstimmt, für das solche Messungen zur Verfügung standen. Zudem konnten wir unsere Entdeckung anhand von 26 experimentellen Studien validieren.
Der Weg dorthin war mit viel Arbeit verbunden. Eine unserer größten Herausforderungen bestand nicht darin, die Komplexität zu erhöhen, sondern herauszufinden, was wir weglassen konnten. Wir hatten uns ehrgeizige Ziele gesetzt: mehrere visuelle Aufgaben modellieren, komplexe Synapsen-Dynamiken implementieren und zahlreiche Vergleiche durchführen. Der Durchbruch gelang uns jedoch erst, als wir die Komplexität unseres Modells sorgfältig reduzierten und uns mit den Unsicherheiten beschäftigten, die sich aus der unvollkommenen Simulation und Optimierung ergeben. Indem wir uns auf die wesentlichen, evidenzbasierten Aufgaben wie die Erkennung des optischen Flusses, ein einfaches Synapsenmodell und die systematische Analyse geeigneter Vergleichsgruppen konzentrierten – immer ein Schritt nach dem anderen -, konnten wir so das Beste aus den Konnektomdaten herausholen.
Wie geht es weiter?
Unser Modell macht Vorhersagen über zahlreiche Aspekte der Informationsverarbeitung im Fliegengehirn. Trotzdem simuliert unser Modell viele biologische Details noch nicht, wie zum Beispiel die Auswirkungen der Neuromodulation, der Gliazellen und der elektrischen Synapsen. Wir stehen noch am Anfang, und es gibt vieles im Nervensystem, das wir zwar messen, aber noch nicht modellieren können. Unser Vorgehen eröffnet neue Möglichkeiten, Modelle iterativ zu verfeinern. Mit der Zeit werden wir in der Lage sein, mehr Messungen zu integrieren, um einerseits unser Wissen über das System und die Modellierungstechniken schrittweise zu erweitern und andererseits Modelle zu erstellen, die sich dem biologischen Gehirn annähern.
Unser Modell erweitert den Werkzeugkasten, der für die Untersuchung von Fruchtfliegen zur Verfügung steht, geht aber darüber hinaus. Unser Ansatz bildet die Grundlage, um mit Hilfe von Konnektomen und maschinellem Lernen herauszufinden, wie biologische neuronale Netzwerke Informationen effizient verarbeiten. Um den Forschungsprozess zu beschleunigen und experimentell überprüfbare Vorhersagen anzuregen, haben wir unsere Werkzeuge der Forschungscommunity zur Verfügung gestellt. So können auch andere Forschende unser Modell für ihre Berechnungen nutzen.
Konnektom-basierte Modelle sind kompakte künstliche Intelligenzen, die biologisch plausibel sind und ihren effizienten biologischen Pendants entsprechen. Mit unserem Ansatz haben wir eine Brücke zwischen künstlichen und biologischen neuronalen Netzen geschlagen, die neue Wege zum Verständnis beider eröffnet.
[1] Llama 3.1 70B’s training: 7.0 million H100-80GB hours at 700W per GPU: 7,000,000 hours × 0.7 kW = 4,900,000 kWh; An average U.S. household uses 1.22 kWh per hour: 4,900,000 kWh ÷ 1.22 kWh/household-hour ≈ 4,016,393 household-hours
Originalpublikation:
Janne K. Lappalainen, Fabian D. Tschopp, Sridhama Prakhya, Mason McGill, Aljoscha Nern, Kazunori Shinomiya, Shinya Takemura, Eyal Gruntman, Jakob H. Macke, Srinivas C. Turaga. Connectome-constrained networks predict neural activity across the fly visual system. Nature 634, 1132–1140 (2024). https://doi.org/10.1038/s41586-024-07939-3
Titelillustration: Franz-Georg Stämmele
Übersetzung ins Deutsche: Fortuna Communication
Von Menschen gesteuerte neuronale Netze für Synchrotron-Experimente


Kommentare